Survival analyse: Difference between revisions
No edit summary |
No edit summary |
||
| Line 3: | Line 3: | ||
|coauthor= dr. R.B. Geskus | |coauthor= dr. R.B. Geskus | ||
}} | }} | ||
Survival analyse wordt gebruikt voor data die informatie geeft over de tijd tot het optreden van een bepaald event. Met tijd wordt in deze bedoeld het aantal jaren, maanden of weken vanaf de start van de follow-up van een patiënt tot aan het optreden van een event. Het event kan overlijden zijn (vandaar de naam survival analyse), maar ook een relapse, herstel of een ander helder gedefinieerd eindpunt. In het algemeen wordt er bij survival analyse vanuit gegaan dat er maar één type risico is en dat een event per individu maar hoogstens één keer kan optreden. Bij meerdere risico's of meerdere herhalingen van een event zijn meer geavanceerde analyse technieken nodig zoals | Survival analyse wordt gebruikt voor data die informatie geeft over de tijd tot het optreden van een bepaald event. Met tijd wordt in deze bedoeld het aantal jaren, maanden of weken vanaf de start van de follow-up van een patiënt tot aan het optreden van een event. Het event kan overlijden zijn (vandaar de naam survival analyse), maar ook een relapse, herstel of een ander helder gedefinieerd eindpunt. In het algemeen wordt er bij survival analyse vanuit gegaan dat er maar één type risico is en dat een event per individu maar hoogstens één keer kan optreden. Bij meerdere/concurrerende risico's of meerdere herhalingen van een event zijn meer geavanceerde analyse technieken nodig zoals recurrent event analyse of [[Survival_analyse#Bronnen_met_betrekking_tot_concurrerende_risico's | competing risk analyse]]. | ||
Bij het meten van tijd-tot-event data is vaak sprake van gecensureerde waarnemingen. Hierbij heb je iets aan informatie over de 'survival time' van een patiënt, maar weet je niet precies hoe lang de tijd-tot-event is. Een voorbeeld. Stel dat in een studie patiënten over een periode van twaalf weken gevolgd worden. Censurering (Eng.: censoring) kan op drie manieren optreden: | Bij het meten van tijd-tot-event data is vaak sprake van gecensureerde waarnemingen. Hierbij heb je iets aan informatie over de 'survival time' van een patiënt, maar weet je niet precies hoe lang de tijd-tot-event is. Een voorbeeld. Stel dat in een studie patiënten over een periode van twaalf weken gevolgd worden. Censurering (Eng.: censoring) kan op drie manieren optreden: | ||
| Line 10: | Line 10: | ||
*Een patiënt trekt zich terug uit de studie na 6 weken, dus voor het eind van de studieperiode. Van deze persoon weten we alleen dat de tijd-tot event langer is dan 6 weken. Of een patiënt start aan de studie maar raakt na het laatste contact op 8 weken 'lost to follow-up'. Ook van deze patiënt weten we alleen dat de eventvrije periode langer is dan 8 weken. | *Een patiënt trekt zich terug uit de studie na 6 weken, dus voor het eind van de studieperiode. Van deze persoon weten we alleen dat de tijd-tot event langer is dan 6 weken. Of een patiënt start aan de studie maar raakt na het laatste contact op 8 weken 'lost to follow-up'. Ook van deze patiënt weten we alleen dat de eventvrije periode langer is dan 8 weken. | ||
*Een patiënt kan niet tot het einde gevolgd worden doordat hij een ander type event krijgt. Bijvoorbeeld: we onderzoeken tijd tot relapse na operatie, maar een patiënt overlijdt tijdens de studie zonder een relapse gehad te hebben. | *Een patiënt kan niet tot het einde gevolgd worden doordat hij een ander type event krijgt. Bijvoorbeeld: we onderzoeken tijd tot relapse na operatie, maar een patiënt overlijdt tijdens de studie zonder een relapse gehad te hebben. | ||
Latest revision as of 13:28, 20 November 2024
| Auteur | dr. ir. N van Geloven | |
| Co-Auteur | dr. R.B. Geskus | |
| auteurschap op deze site | ||
Survival analyse wordt gebruikt voor data die informatie geeft over de tijd tot het optreden van een bepaald event. Met tijd wordt in deze bedoeld het aantal jaren, maanden of weken vanaf de start van de follow-up van een patiënt tot aan het optreden van een event. Het event kan overlijden zijn (vandaar de naam survival analyse), maar ook een relapse, herstel of een ander helder gedefinieerd eindpunt. In het algemeen wordt er bij survival analyse vanuit gegaan dat er maar één type risico is en dat een event per individu maar hoogstens één keer kan optreden. Bij meerdere/concurrerende risico's of meerdere herhalingen van een event zijn meer geavanceerde analyse technieken nodig zoals recurrent event analyse of competing risk analyse.
Bij het meten van tijd-tot-event data is vaak sprake van gecensureerde waarnemingen. Hierbij heb je iets aan informatie over de 'survival time' van een patiënt, maar weet je niet precies hoe lang de tijd-tot-event is. Een voorbeeld. Stel dat in een studie patiënten over een periode van twaalf weken gevolgd worden. Censurering (Eng.: censoring) kan op drie manieren optreden:
- Een patiënt is gevolgd vanaf het begin van de studie tot aan het eind van de 12-weekse studieperiode. Op dat moment heeft de patiënt het event nog niet gekregen. We weten nu alleen dat de tijd-tot-event van deze persoon langer is dan 12 weken.
- Een patiënt trekt zich terug uit de studie na 6 weken, dus voor het eind van de studieperiode. Van deze persoon weten we alleen dat de tijd-tot event langer is dan 6 weken. Of een patiënt start aan de studie maar raakt na het laatste contact op 8 weken 'lost to follow-up'. Ook van deze patiënt weten we alleen dat de eventvrije periode langer is dan 8 weken.
- Een patiënt kan niet tot het einde gevolgd worden doordat hij een ander type event krijgt. Bijvoorbeeld: we onderzoeken tijd tot relapse na operatie, maar een patiënt overlijdt tijdens de studie zonder een relapse gehad te hebben.
Kaplan-Meier-analyse
Wanneer gebruik ik een Kaplan-Meier-analyse?
Om de tijd tot het optreden van het event voor de gehele steekproef te kwantificeren, wordt er vaak een Kaplan-Meier-analyse gebruikt. De Kaplan-Meier-analyse is een niet-parametrische aanpak. Dat wil zeggen dat er geen aanname gedaan wordt ten aanzien van het soort verdeling (normaal, gamma etc.) van de event tijden. Toch zitten er wel een paar aannames achter deze methode:
- De prognose van patiënten die vroeg of laat zijn geïncludeerd in de studie is gelijk.
- Patiënten die gecensureerd zijn op een bepaald tijdstip hebben dezelfde prognose als patiënten die op datzelfde tijdstip wel in de studie zijn gebleven (en op dat moment nog geen event hebben gehad). Er wordt niet aan deze aanname voldaan als bijvoorbeeld personen met een grote kans op een event vlak voordat het event optreedt de studie verlaten.
- De tijdstippen waarop de events optreden zijn exact bekend.
- Patiënten worden vanaf het begin van de onderzochte periode gevolgd. Als er bijvoorbeeld pas 10 dagen na interventie gestart wordt met het volgen van survival status van patiënten, dan zullen patiënten die in de eerste 10 dagen reeds overleden zijn niet vertegenwoordigd zijn in de studie. Een Kaplan-Meier-analyse geeft dan geen goede inschatting van de echte survival tijd. Survival modellen die rekening houden met dit verschijnsel worden late entry survival modellen genoemd.
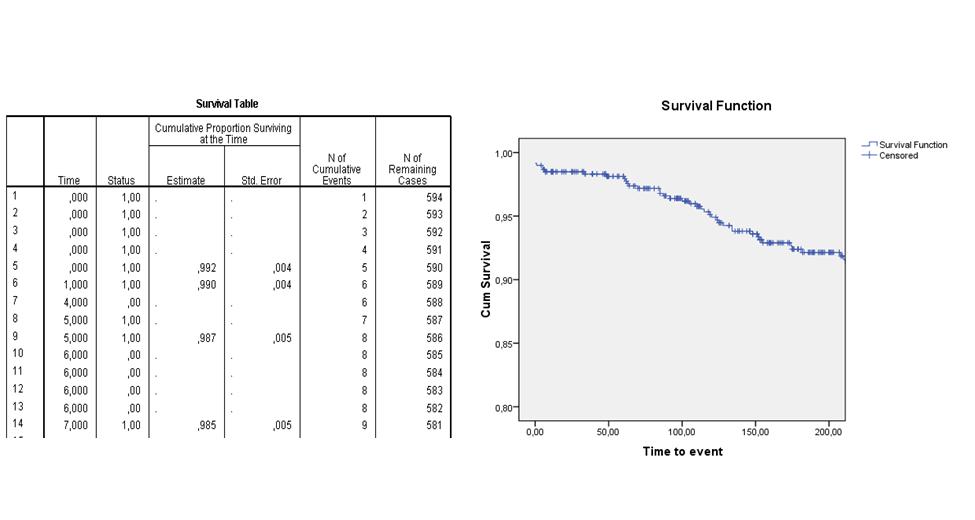
De basisgedachte achter de Kaplan-Meierprocedure is het opdelen van de tijdsschaal. Op ieder moment dat er bij één of meerdere patiënten een event optreedt, wordt de kans daarop geschat door het aantal events op dat moment te delen door het aantal personen dat op dat moment nog in de studie zit (i.e. niet eerder al een event heeft gehad of gecensureerd is). De survival functie (of cumulatieve proportie van 'survivors') combineert deze schattingen over alle tijdstippen. Op deze manier wordt het resultaat minimaal beïnvloed door patiënten die halverwege de rit gecensureerd raken. Een Kaplan-Meier-analyse resulteert in een 'life table' en een 'survival curve'.
De volgende life table en survival curve laten bijvoorbeeld zien wat de verwachte overlevingskans is van patiënten die een dotterbehandeling hebben ondergaan. Je ziet in de tweede kolom ("Time") van de life table dat er op dag 4 en dag 6 patiënten gecensureerd raakten en dat er op dag 0 vijf events waren, op dag 1 een event, op dag 5 twee events en dag 7 een event optrad. Op de dagen dat er een event optrad, is de cumulatieve survival berekend. In de survival curve zie je de geschatte survival functie over de eerste 200 dagen na interventie. Na 200 dagen wordt de kans op overleven geschat op ongeveer 92%.
Hoe test ik of Kaplan-Meier survival curves van elkaar verschillen?
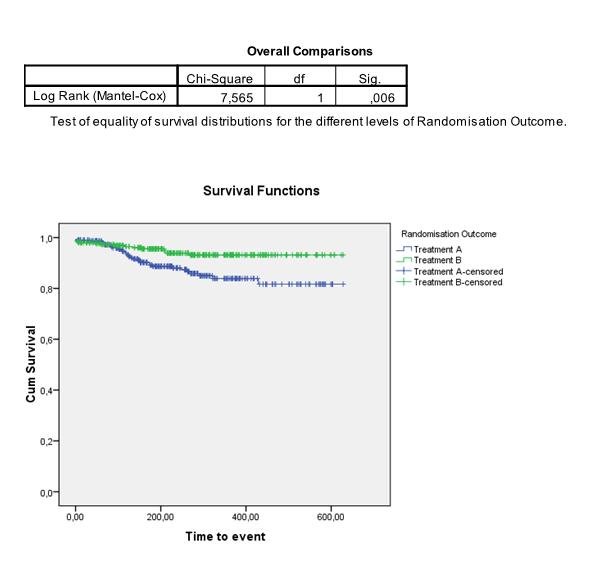
We kunnen de curves ook apart schatten en vergelijken voor twee (of meer) subgroepen in onze data. Om te testen of de Kaplan-Meier curves van 2 (of meer) groepen significant van elkaar verschillen, wordt vaak de log-rank test gebruikt. Hierbij is de nulhypothese dat de curves niet van elkaar verschillen. De test vergelijkt de geobserveerde verschillen tussen de survival curves met verschillen die zouden kunnen ontstaan als er geen verschil is tussen de groepen. In het volgende voorbeeld concluderen we bijvoorbeeld dat het verschil tussen treatment A en treatment B significant is:
Mijn Kaplan-Meier curves kruisen, mag ik dan nog de log-rank test gebruiken?
Bij mijn Kaplan-Meier-analyses voor het vergelijken van een factor, heb ik curves die elkaar kruisen. In statistische stukken wordt soms gezegd dat een log-rank test dan niet geschikt is om een potentieel verschil tussen curves te analyseren. Desondanks, als ik met SPSS een log-rank test uitvoer, krijg ik soms alsnog een significante p-value. Houdt de log-rank test (als uitgevoerd door SPSS) rekening met het feit dat de curves elkaar kruisen? Of zou ik eigenlijk geen log-rank mogen gebruiken dan?
De log-rank test houdt geen rekening met het kruisen van curves. De test bekijkt het verschil tussen de curves op ieder (event-)tijdspunt en telt de gevonden verschillen (in detail het verschil tussen observed en expected events) op over alle tijdspunten. Als 2 curves bijvoorbeeld in het midden kruisen, dan zal in de vroege periode de ene groep relatief meer events hebben en in de late periode de andere groep. Bij het optellen van de verschillen zullen deze twee periodes elkaar 'uitmiddelen'. Grote kans dat de log-rank statistic dan niet significant is, terwijl er toch interessante verschillen kunnen zijn in de twee periodes apart. Om deze reden wordt de log-rank test als minder geschikt (eigenlijk minder krachtig, minder power) gezien bij kruisende curves.
In jouw geval waarbij je kruisende curves hebt en toch een significante log-rank test, zal het verschil in een van de twee periodes (voor of na het omslagpunt van de hazard) sterker zijn dan in de andere periode. Er is dan toch 'netto' een verschil 1 kant op gevonden. Dit kan informatief zijn, al moet je goed oppassen hoe zo'n verschil te interpreteren. Zie ook deze ingezonden brief n.a.v. van een verkeerde interpretatie in zo'n situatie.
Ik raad je aan om in ieder geval de plot van de survival curves mede te rapporteren (en niet alleen de log-rank statistic), zodat de lezer zelf kan beoordelen waar het verschil te verwachten is. Statistische alternatieven zijn het gebruik van testen die meer gewicht leggen op vroege of juist late periodes. Bijvoorbeeld de Wilcoxon test (in SPSS de Breslow test genoemd), deze test weegt verschillen in het begin van de survival curves zwaarder dan verschillen aan het einde van de curves. De test is bijvoorbeeld geschikt wanneer het effect van een behandeling op survival het sterkst is in de eerste fase en later minder effectief wordt en je wilt inzoomen op de eerste fase. Of de Tarone-Ware test. Deze test geeft net als de Wilcoxon test meer relatief meer gewicht aan verschillen op vroege momenten (maar minder uitgesproken dan de Wilcoxon test). Ook kun je overwegen de analyse apart te doen over twee tijdsintervallen, al is het dan heel lastig om te bepalen waar je de grens trekt. Het liefst zou je dergelijke alternatieve analyses al vooraf hebben gespecificeerd.
Ik heb veel gecensureerde patiënten, kan ik de Breslow of Tarone-Ware test gebruiken in plaats van de log-rank test?
De test die ik nu gebruik is logrank. Ik zag dat er ook 2 andere testen zijn (Breslow en Tarone-Ware). Het is me niet helemaal duidelijk wanneer je deze gebruikt behalve de vernoeming dat deze toetsen vooral het begin van de curve beoordelen. De reden dat ik mogelijk een andere test wil proberen is het feit dat ik veel censored zie in de curve. De patiënten zijn niet allemaal even lang gevolgd en dus neemt het aantal patiënten in follow up af naarmate de tijd verstrijkt. Kan/mag de Breslow of Tarone-Ware in dit geval?
De Breslow en Tarone-Ware 'luisteren' inderdaad meer naar de beginperiode wanneer er nog meer personen in follow up zijn dan naar de eindperiode. Echter, het feit dat er later in de follow up veel gecensureerd wordt, is niet een reden om voor deze testen te kiezen. Ook de log-rank test zal namelijk het meest gevoed worden door de periodes waar er de meeste mensen nog in follow up zijn. De Breslow / Tarone-Ware zijn aan te raden wanneer er op basis van een biologische hypothese de verwachting is dat er in de beginperiode de grootste verschillen zullen zijn (bijvoorbeeld bij een eenmalige behandeling waarbij het effect uitvaagt over de tijd). Het zijn niet de standaardtesten, dus het gebruik ervan zal duidelijk gemotiveerd moeten worden. Ik raad dus aan hier de 'gewone' log-rank te gebruiken, de latere momenten met minder personen zullen vanzelf minder zwaar meetellen in de analyse.
Kan ik meerdere aparte Kaplan-Meier curves bij elkaar in 1 grafiek plotten?
Voor een paper heb ik 3 Kaplan-Meier curves die op de x-as allemaal even ver lopen (allemaal een tijdsduur van 27 maanden). Ik wil deze 3 curves in 1 plot zetten. Kan dat met SPSS en zo ja, hoe?
We zijn er zelf uitgekomen en wel op de volgende manier: In ons geval wilden wij 3 verschillende curves (survival, grote amputatie en kleine amputatie) in 1 KM grafiek zetten. We hebben de data opnieuw gerangschikt. 1 variable met time to event, 1 variable met event status (alledrie de events hadden status 1), en een nieuwe onderscheidende variabele die duidelijk maakte dat de eerste time-to-event en event zelf sloeg op survival, tweede time-to-event en event zelf op grote amputatie en derde time-to-event en event zelf op kleine amputatie. Invoeren gewoon time-to-event en event status en de nieuwe onderscheidende variabele wordt de 'factor'. Mocht iemand anders ooit nog met hetzelfde probleem zitten, bij deze de oplossing.
Hoe kan ik mijn logrank analyse doen over alleen het eerste jaar van de follow up?
Op een polikliniek voor HIV-geïnfecteerde kinderen in Malawi hebben we in de periode van 2004-2008 een cohort kinderen gevolgd vanaf het moment dat zij startten met antiretrivirale therapie. We zijn geïnteresseerd in hun uitkomsten en risicofactoren voor mortaliteit en uitval uit behandeling (LTFU). 1) Ik probeer uitkomsten na 12 maanden op een rij te zetten d.m.v. analyze_survival_lifetables. Dit lijk te lukken. 2) Ik wil naar voorspellende factoren kijken voor overlijden en dood op 12 maanden. Voor univariate analyses zou ik hiervoor willen gebruiken: analyze_survival -> kaplanmeier met compare factor log-rank test. Hiermee doe ik een analyse voor uitkomst na totale duur follow up. Het lukt mij niet hierbij specifiek naar uitkomsten tot 12 maanden te kijken.
Als er een langere follow-up is, en je wilt in een analyse alleen focussen op de periode tot 12 maanden, dan is het nodig om de informatie van de personen op 12 maanden te censureren. Als het goed is heb je nu in de dataset de volgende 2 variabelen:
-tijd tot laatste follow-up of tijd tot optreden event (mortaliteit of uitval behandeling): FUtime
-event indicator (wel of niet het event opgetreden): FUstatus (1=event, 0= geen event)
Om alleen op de eerste 12 maanden te focussen, is het nodig om de volgende aanpassingen te maken:
indien FUtime>12 en FUstatus =0 -> FUtime=12 FUstatus=0
indien FUtime>12 en FUstatus =1 -> FUtime=12 FUstatus=0
indien FUtime=<12 en FUstatus =1 -> variabelen blijven hetzelfde
indien FUtime=<12 en FUstatus =0 -> variabelen blijven hetzelfde
Met de nieuwe FUtime (die nooit groter is dan 12) en de nieuwe status (die nu iets minder events bevat) kan een Kaplan-Meier-analyse gedaan worden voor alleen het eerste jaar.
Hoe ga ik om met patiënten die vanwege overlijden niet meer het event van interesse kunnen krijgen?
Ik heb een RCT gedaan met een follow-up van 1 jaar met als belangrijkste uitkomstmaat: het aantal dagen vanaf randomisatie tot terugkeer naar werk. Nu wil ik onderzoeken of de tijd tot terugkeer naar werk significant verschilt tussen beide groepen (controlegroep en interventiegroep). Hiervoor heb ik KM-survival analyse gedaan met logrank toets. Nu is het zo dat een aantal patiënten gedurende de follow-up periode overleden zijn en een aantal patiënten palliatief worden behandeld. Deze patiënten kunnen de positieve uitkomstmaat (weer aan het werk) nooit meer bereiken. Hoe ga je om met deze patiënten in de survival analyse? Moet ik deze patiënten helemaal uit de analyse halen? Moet ik deze als censored beschouwen? Moet ik deze patiënten zien als negatieve uitkomst (= niet aan het werk). Kortom wat is het effect van censored data op een survival analyse en logrank toets en welke afweging is het meest juist?
De voor- en nadelen van de verschillende alternatieven:
1. de overleden/palliatieve patiënten in de KM-analyse laten en censureren op het moment van dit event
Voordeel: de informatie van deze patiënten voordat ze overleden / voordat ze palliatief waren doet mee in de analyse / geen verlies van data Nadeel: de KM-analyse veronderstelt dat de prognose van deze patiënten, nadat ze gecensureerd worden, gelijk is aan patiënten die nog wel in de analyse zitten (aanname van non-informative censoring). De schattingen (en vergelijkingen) op latere follow up momenten worden daardoor (onevenredig) veel bepaald door relatief goede patiënten die wellicht niet representatief zijn voor de totale groep.
2. de overleden/palliatieve patiënten in de KM-analyse uit de analyse laten
Voordeel: eenvoudig uit te leggen aanpak, je beperkt de studiegroep alsof het een exclusiecriterium is Nadeel: alleen als op t=0 al bekend is dat iemand tot de 'exclusiegroep' behoort is dit een logische aanpak, voor deze personen zou je kunnen zeggen dat de interventie toch geen invloed meer kan hebben op terugkeer naar werk omdat dat (ongeacht welke behandeling ook) geen resultaat hierop kan hebben. Voor personen die in het begin nog alle kanten op kunnen en pas later achteruit gaan zou het zonde zijn/kan het vertekening geven die info niet te gebruiken.
3. de overleden/palliatieve patiënten in de KM-analyse als negatieve uitkomst zien / Competing risk analyse
Voordeel: Je zou kunnen ageren dat deze patiënten een ultiem 'slechte' uitkomst hebben en ook als zodanig meegerekend moeten worden. Je zegt dan eigenlijk dat ze het event 'terug naar werk' nooit meer zullen bereiken. Hun status staat dan op 0, en de follow up tijd zou je (gekunsteld) op oneindig lang kunnen zetten (of in ieder geval langer dan de langste follow up). Je kunt dit ook formeel in een competing risk analyse doen. Nadeel: de analyse is minder standaard / competing risk analyse wordt niet standaard in SPSS aangeboden.
Hoe bereken ik de 'events per person year'?
Ik wil graag mijn uitkomsten (opnames in verband met verklevingen in de buik en het vóórkomen van littekenbreuken) weergeven als 'event per person-years at risk', om te corrigeren voor de wisselende follow up duur. Hoe kan ik dit het beste doen? Ik zou een Kaplan-Meier kunnen maken van time-to-event, maar ik wil ook graag deze maat weergeven. Moet ik dan een variabele aanmaken waarin ik event ja/ nee deel door de tijd 'at risk'? Of is er een functie in SPSS waarmee ik dit makkelijker kan berekenen?
De uitkomst 'events per person-years at risk' is simpel te berekenen door alle events bij elkaar op te tellen (som_events), alle tijd at risk bij elkaar op te tellen (som_tijd) en die twee op elkaar te delen: som_events/som_tijd.
Omdat SPSS gewend is om te rekenen 'per rij' en niet zozeer per kolom, zijn deze optellingen makkelijker te doen als je de data even in een ander pakket (excel of R) zet.
Bij de maat 'events per person-years at risk' wil je waarschijnlijk ook een betrouwbaarheidsinterval berekenen? Die zou je kunnen berekenen m.b.v. de calculator op www.openepi.com. Kies in de linkerbalk voor person time -> 1 rate. Geef het aantal events en de totale tijd in. Je krijgt dan de rate en een betrouwbaarheidsinterval terug. Let goed op, de rate wordt per 10, 100, 1000 person years gegeven (afhankelijk van het aantal informatieve decimalen). Het is dus goed om de getallen naast je eigen berekening (zoals boven) te leggen.
Waar vind ik de Kaplan-Meier-analyse in SPSS?
Je vindt de Kaplan-Meier-analyse in SPSS 28 onder Analyze -> Survival -> Kaplan-Meier.
Cox-regressie
Wanneer gebruik ik een Cox proportional hazards model?
Wanneer er bij een survivalanalyse slechts één voorspellende factor onderzocht wordt die slechts een paar mogelijke waardes kan aannemen, kan een Kaplan Meier analyse volstaan. Zijn er echter meerdere factoren die de overleving kunnen beïnvloeden, dan is een uitgebreider model nodig. Een veel gebruikt model is het Cox proportional hazards model. Hiermee kun je net als met andere multivariabele regressie modellen meerdere voorspellers tegelijk bestuderen. Voordeel van het Cox-model boven een Kaplan-Meier-analyse is bovendien dat er naast de vraag "is er een effect" ook antwoord gegeven wordt op de vraag "hoe groot is het effect" van een bepaalde voorspeller. Het Cox-model geeft de grootte van een effect weer met behulp van hazard ratio's (HR), waarbij een HR van 1 aangeeft dat er geen verschil is. De hazard is de instantane kans om een event te krijgen, en een hazard ratio is vergelijkbaar met een relatief risico. Bijvoorbeeld: "During the 20-year follow-up, 83 women developed lung cancer. There was a moderate association between hormone use and lung cancer (hazard ratio, 1.13; 95% CI, 1.08-1.73)."
Het Cox-model maakt een belangrijke aanname: de proportional hazards assumptie. Deze zegt dat de HR's constant zijn in de tijd. Dit betekent dat het relatieve verschil in hazard veroorzaakt door verschillende waardes van een voorspeller gelijk blijft in de tijd. Er wordt in het bovenstaande voorbeeld dus vanuit gegaan dat het wel of niet gebruiken van hormonen een zelfde invloed heeft na 1 jaar als na 20 jaar follow up: op ieder tijdstip wordt de kans op het optreden van longkanker bij hormoongebruik 1.13 keer hoger geschat dan dezelfde kans als er geen hormonen werden gebruikt. Bij een Cox-regressie, zijn er verder geen specifieke aannames over de verdeling van de hazard functie of voorspellende variabelen. Ze hoeven dus niet normaal verdeeld te zijn .
Hoe test ik de proportional hazards-aanname binnen een Cox-model?
Er zijn meerdere manieren om de PH-aanname van een Cox-model te testen:
- Grafisch
Als een variabele in het model voldoet aan de proportional hazards (PH) aanname, dan zullen de geschatte overlevingscurves, op de zogenaamde log-schaal, voor de waardes van deze variabele parallel lopen. Je kunt hier naar kijken door in het Cox-model de variabele voor welke je de PH-aanname wilt onderzoeken niet meer als covariaat, maar als stratum mee te geven en te vragen om log-minus-log survival plots. De resulterende lijnen moeten op het oog parallel lopen (zeker elkaar niet kruisen) om te voldoen aan de PH-aanname voor deze variabele. Voor iedere variabele in het model is een dergelijke plot nodig. In deze voorbeeldsyntax kun je zien hoe het maken van een log-minus-log-plot er in SPSS syntax uit komt te zien.
- Testen van (Schoenfeld-) residuen
Er kan een formele test gedaan worden m.b.v. Schoenfeldresiduen. Er is geen standaardfunctie hiervoor in SPSS. In Stata (functie estat phtest) of R (functie cox.zph) kan het wel direct.
- Toevoegen van een tijdsafhankelijke variabele
Anticiperend op het mogelijk niet voldoen aan de PH-aanname kun je ook een tijdsafhankelijk Cox-model draaien met daarin een tijdsafhankelijke variabele opgenomen (bijvoorbeeld GENDER*T). Als deze tijdsafhankelijke variabele significant bijdraagt aan het model, is er blijkbaar niet aan de PH-aanname voldaan.
Mijn variabelen lijken niet te voldoen aan de proportional hazards-aanname, mag ik ze nu niet meenemen in de Cox-regressie?
Ik ben aan de slag gegaan met het testen van de proportional hazards-aanname. In de log-log-plots kruisen de lijnen van bijna alle covariaten. Betekent dit dat ik al deze covariaten niet mee mag nemen in de analyse?
Het is niet zo dat als de lijnen kruisen dat je de variabele dan sowieso niet mee mag nemen. Je moet per variabele bekijken wat er aan de hand is. Mogelijk opties:
1. Een variabele is niet gerelateerd aan de uitkomstmaat. De log-minus-log survival curves lopen dan naar verwachting over elkaar heen. Door random fluctuatie kunnen de curves ook 'om elkaar heen wiebelen' en elkaar dus meerdere keren kruisen. Conclusie is hier dat er geen effect is (ook geen proportioneel effect).
2. Een variabele is gerelateerd aan de uitkomstmaat, maar de curves kruisen op een gegeven ogenblik. Hier is het lastiger. Als de curves kruisen, dan is het effect van een variabele niet proportioneel. Dat wil zeggen, op het ene tijdsinterval is de ene groep beter af en op het andere tijdsinterval de andere groep. Als de Cox-analyse toch een proportioneel effect van een variabele aanwijst, dan betekent dit zoveel als dat er gemiddeld over de hele tijdslijn toch 1 van de 2 groepen beter uit de bus komt. 'Uitgemiddeld' doet deze groep het dan beter. Dit kan in bepaalde situaties een valide conclusie zijn, maar wanneer alleen de Cox-resultaten (HR's) getoond worden vertelt dit niet het hele verhaal. Bijvoorbeeld als een groep in het begin een groot voordeel laat zien, maar richting het einde van de follow-up het slechter doet dan zijn tegenhanger, dan is het te verwachten dat in een nog langere follow-up-periode het effect om kan gaan slaan. Deze informatie is goed om erbij te vermelden; als een lezer alleen de HR ziet, denkt ie dat de groep het overal in de tijd beter zal doen. Wanneer de variabele duidelijk (en verklaarbaar) een differentieel effect in de tijd laat zijn, dan kan het verstandig te zijn om 2 HR's te schatten, 1 voor de vroege periode en 1 voor de late. Dit kan m.b.v. het aanmaken van een tijdsafhankelijk covariaat.
Hoe kan ik de output lezen van de analyse van een categorische variabele in een Cox-model?
Voor een cohort HIV-geïnfecteerde kinderen, gevolgd vanaf het moment dat ze met antiretrovirale medicatie starten, ben ik op zoek naar voorspellers van overlijden. Ik ben nu bezig met de Cox-regressie (vooralsnog bezig met univariate analyse). Hieronder een voorbeeld van output van een categoriale variabele (leeftijdscategorieën) met meer dan 2 categorieën (referentie = kinderen >12 jaar oud, van wie we verwachten dat ze de beste prognose hebben). (1) (2) (3) betreffen kinderen van 0-1 jaar, 1-5 jaar en 5-12 jaar oud. Wat betekent de p-waarde die genoemd staat op de regel van de referentiewaarde?
| Variables in the Equation | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| B | S.E. | Wald | df | Sig. | Exp(B) | 95.0% C.I.for Exp(B) | |||
| Lower | Upper | ||||||||
| Leeftijd | leeftijd | 23.539 | 3 | .000 | |||||
| leeftijd(1) | 1.094 | .261 | 17.613 | 1 | .000 | 2.985 | 1.791 | 4.795 | |
| leeftijd(2) | .773 | .211 | 13.379 | 1 | .000 | 2.166 | 1.431 | 3.276 | |
| leeftijd(3) | .351 | .236 | 2.206 | 1 | .137 | 1.420 | .894 | 2.256 | |
De eerste regel laat het resultaat zien van de overall-test voor de variabele. Dit is de test die als nulhypothese heeft dat alle 4 de categorieën dezelfde kans (eigenlijk hazard) hebben op overlijden. De toets kijkt of er ergens een verschil is in categorieën, je ziet ook dat de test 3 'degrees of freedom' heeft. Je kunt het vergelijken met een ANOVA test bij het vergelijken van gemiddelde waarden in 4 groepen. De test hoort dus niet specifiek bij de referentiecategorie, dat vindt SPSS gewoon een handige locatie om 'm neer te zetten. In principe ga je als volgt te werk: je kijkt eerst of de overall test significant is. Als dat zo is, dan ben je geoorloofd om door te gaan en te kijken naar de verschillen tussen de categorieën onderling.
Hoe rapporteer ik de resultaten van een categorische variabele in een Cox-model?
Ik heb een Cox-regressie uitgevoerd met een dichotome uitkomstmaat (wel/geen ontwikkeling van artritis) en de variabele is een categorische maat (0-4). Normaal gesproken ben ik gewend om de uitslag met een continue variabele als volgt te noteren: HR x.x (95% CI x.x to x.x); p=0.xxx. Nu las ik echter dat je vanwege de categorische maat moet kijken naar de Wald-uitslag; hier komt wel een p-waarde uit, maar geen CI e.d. Hoe moet je deze uitslag dan noteren in een manuscript?
Meestal worden de significantietoetsen van HR's in Cox-regressie met een Wald statistic gegenereerd. Dat geldt evengoed voor continue als voor categorische voorspellers. Wat hier wellicht het issue is, is dat er bij het evalueren van een categorische voorspeller eerst bekeken moet worden of de 'overall' toets significant is, dat wil zeggen of de toets die kijkt of het opsplitsen in de 5 categorieën (met 4 df) bijdraagt aan het model significant is. Als die uitslag positief is, kun je door gaan met inzoomen op de verschillende contrasten (HR's) apart en deze op gebruikelijke wijze met 95% CI rapporteren. De p-waardes van de losse HR's zijn doorgaans wat minder relevant omdat deze zeer afhankelijk zijn van de gekozen referentiecategorie.
Mijn Cox-regressie convergeert niet, heb ik wel de juiste analyse gebruikt?
Ik wil graag een Cox-regressie doen op een patiëntencohort om te bepalen of sommige variabelen een independent predictive value hebben. Ik heb een cohort van 86 patiënten. Voor de cohort wil ik 8 variabelen vergelijken, 7 binaire en een continue (leeftijd). In de cohort waren er 6 events. Als ik het Cox regression model dan probeer te runnen krijg ik de volgende warning: ‘Since coefficients did not converge no further models will be fitted’. De tabel geeft dan voor alle variabelen een significantie van 1 en een hazard ratio van 1. Wat betekent dit? En is een Cox regression wel de geschikte test voor mijn data dan?
Ik denk dat je last hebt van een ernstige vorm van te weinig power. Je hebt maar 6 events in de data. Voor een Cox-regressie zijn er volgens de vuistregels 10 a 20 events per parameter die je wilt testen nodig. Met 6 events kun je helaas niet zinvol naar effecten van variabelen kijken m.b.v. een Cox-model. Uiteraard kun je wel de ruwe data beschrijven.
Waar vind ik het Cox proportional hazards model in SPSS?
Je vindt deze in SPSS 28 onder Analyze -> Survival -> Cox Regression of Cox w/ Time-Dep Cov.
Bronnen met betrekking tot concurrerende risico's
- www.competingrisks.org “Data Analysis with Competing Risks and Intermediate States” by Ronald B. Geskus. Forthcoming July 2015 by Chapman and Hall/CRC, ISBN 9781466570351
- Competing risk analysis using R: an easy guide for clinicians - Technical Report, L Scrucca, A Santucci and F Aversa, Bone Marrow Transplantation (2007) 40, 381-387
- Concurrerende risico's in klinisch onderzoek, Rob C.M. van Kruijsdijk, Marinus J.C. Eijkemans en Frank L.J. Visseren, Ned Tijdschr Geneeskd. 2012;156:A5176
- Verduijn M, Grootendorst DC, Dekker FW, Jager KJ, le Cessie S. The analysis of competing events like cause-specific mortality--beware of the Kaplan-Meier method. Nephrol Dial Transplant. 2011 Jan;26(1):56-61. doi: 10.1093/ndt/gfq661
Aanvullende bronnen
- Altman DG, Bland JM. Time to event (survival) data. BMJ 1998;317:468-9.
- Bewick V, Cheek L, Ball J. Statistics review 12: Survival analysis. Critical Care 2004, 8:389-394.
- Bland JM, Altman DG. Survival probabilities. The Kaplan-Meier method. BMJ 1998;317:1572.
- Bland JM and Altman DG. The log rank test. BMJ 2004;Vol 328 may 2004.
- An Introduction to Survival Analysis - by Mark Stevenson from EpiCentre, IVABS, Massey University. 31 pagina's cursusmateriaal (Engels) met voorbeeldsyntax in R.
- Singh R and Mukhopadhyay K. Survival analysis in clinical trials: Basics and must know areas. Perspect Clin Res. 2011 Oct-Dec; 2(4): 145–148.
- Hosmer, D. W., and S. Lemeshow. 1999. Applied Survival Analysis. New York: John Wiley and Sons.
- Kleinbaum, D. G. 1996. Survival Analysis: A Self-Learning Text. New York: Springer-Verlag.
- Norusis, M. 2004. SPSS 13.0 Advanced Statistical Procedures Companion. Upper Saddle-River, N.J.: Prentice Hall, Inc..
- R tutorial survival analyse - Youtube-video met uitleg over uitvoeren van een survivalanalyse in R.
- R tutorial Cox analyse - Youtube-video met uitleg over uitvoeren van een Cox-analyse in R.
Klaar met lezen? Je kunt naar het OVERZICHT van alle statistische onderwerpen op deze wiki gaan of naar de pagina KEUZE TOETS voor hulp bij het uitzoeken van een geschikte toets of analyse. Wil je meer leren over biostatistiek? Volg dan de AMC e-learning Practical Biostatistics. Vind je op deze pagina's iets dat niet klopt? Werkt een link niet? Of wil je bijdragen aan de wiki? Neem dan contact met ons op.
De wiki biostatistiek is een initiatief van de voormalige helpdesk statistiek van Amsterdam UMC, locatie AMC. Medewerkers van Amsterdam UMC kunnen via intranet ondersteuning aanvragen.